Podstawowa recepta na Big Data w języku R

Wszelkiego rodzaju optymalizacje stanowią bardzo czasochłonną pracę. Są jednak bardzo ważne z punktu widzenia budowania szybkich modeli liczących, uczących się zbiorów danych. Podzielę się moim doświadczeniem w tym zakresie. Artykuł będzie dotyczył podstawowych zagadnień z języka R. Poniżej zestawiam kilka punktów, które znacznie przyśpieszą pracę z dużymi zbiorami danych.
Mariusz Kupczyk. Absolwent Uniwersytetu im. Adama Mickiewicza w Poznaniu, na kierunku Geografia, spec. Hydrologia, Meteorologia i Klimatologia. Obecnie pracuje w języku R oraz SAP CRM w firmie Eurocash S.A. Pasjonat analizy i wizualizacji dużych zbiorów danych, w języku programowania “R”. Hobbystycznie pracuje nad automatyzacją przetwarzania danych synoptycznych. Współtwórca strony internetowej związanej z prognozowaniem pogody oraz Twórca systemu automatycznej wizualizacji danych meteorologicznych modelu prognozy pogody WRF – meteoraport.pl.
1. Co można przyspieszyć w kodzie?
2. Jak wykorzystać moc obliczeniową maszyny?
3. Jak zapisywać dane?
Zgodnie z powyższymi punktami, przedstawię kilka wskazówek, szczególnie dla osób początkujących w tematach optymalizacji danych.
Spis treści
Co można przyspieszyć w kodzie?
Temat rzeka, ponieważ tak naprawdę można zrobić bardzo dużo poprawek w tym zakresie. Od zmiany składni języka po dodanie fragmentów kodu z innych języków programowania (np. C++ lub FORTRAN). Język R jest mocno rozbudowany pod tym względem, dlatego warto zapoznać się bardziej z możliwościami integracji różnych języków. Podstawowym problemem wolnego działania są pętle, które w języku R można zastąpić na wiele różnych sposobów. Przedstawię parę istotnych elementów, które według mnie są jasne i proste do zrozumienia.
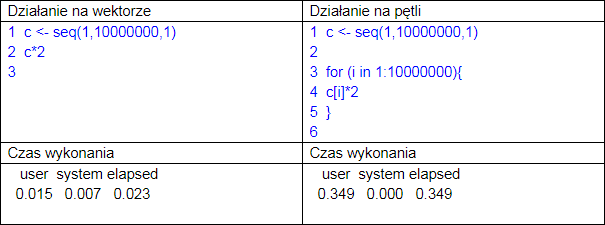
Pierwsze i najbardziej istotne w języku R są operacje na wektorach. Jeśli mamy zbudowany wektor, którego elementy chcemy pomnożyć przez taką samą wartość, nie róbmy tego w pętli! Poniżej zamieszczam podstawową różnicę w kodzie i szybkości działania:
Po lewej stronie mamy działanie na wektorze, którego składnia jest znacznie prostsza od działania pętli. W pierwszym przypadku wystarczy pomnożyć wektor przez stałą liczbę, w drugim, pętla przechodzi kolejno przez dane w wektorze, które mnoży. Czas wykonania widać gołym okiem. Jak radzić sobie z takimi przypadkami?
Po pierwsze zobacz, czy twój kod wygląda przejrzyście, czy może jest mało czytelny i ciężki do zrozumienia. Jeżeli tak, oznacza to prawdopodobnie, że da się go uprościć. Podstawową metodą uproszczenia jest korzystanie z operatorów. W R jest ich kilka (+,-,*, itp.), szczególnie ważny z punktu widzenia filtrowania danych i operacji na danych jest operator uwalniany podczas uruchamiania biblioteki dplyr. Wspomniana biblioteka zawiera zestaw wielu narzędzi, które pozwalają filtrować dane, agregować lub wybierać te, które nas interesują.
Z punktu widzenia programowania, pakiet ułatwia czytanie składni i tworzenie prostych instrukcji dotyczących przetwarzanych zbiorów danych.
Przyjmijmy, że mamy zbiór danych uczniów z klasy maturalnej. Chcemy szybko wyfiltrować dane.
c%>%filter(dane > 85 & klasa == "III")%>%select(Klasa, Nazwisko, Imie)
Wybieramy zbiór klasy (c), filtrujemy dane uczniów, których wyniki są wyższe niż 85% i są to klasy III. W następnym kroku wybieramy interesujące nas dane, czyli klasa, nazwisko i imię uczniów. Tego typu sposób zapisu kodu, jest możliwy dzięki operatorowi %>%. Do szczegółowych informacji dotyczących jego użycia, odsyłam do dokumentacji pakietu dplyr.
Użycie pakietu dplyr, oprócz łatwego programowania zwiększa szybkość działania na danych. Pakiet ten pozwala działać na bardzo dużych zbiorach danych, sprawnie je przetwarzając. Wyobraźmy sobie teraz, że mamy dane z całej Polski, a chcemy uzyskać dostęp do wyników tylko z naszej szkoły. Ile danych należałoby przepuścić przez pętle czy funkcję, zanim uzyskalibyśmy satysfakcjonujący efekt.
Jak wykorzystać moc obliczeniową maszyny?
Duża ilość RAMu, wiele rdzeni, w teorii nasza szybka i często droga maszyna obliczeniowa nie jest wykorzystana. Podstawowa składnia języka R działa standardowo na jednym rdzeniu, przez co mimo 8-u, 16-u lub więcej rdzeni korzystamy tak naprawdę z jednego. Dlaczego nie wykorzystać przetwarzania wielopotokowego? W R na szczęście jest to możliwe i dosyć proste do napisania.
Konieczna jednak będzie biblioteka, która umożliwia działanie na wielu rdzeniach jednocześnie. W artykule pokażę przykład działania biblioteki multicore.
1 for (i in 1:10000000){
2 c[i]*2
3 }
4
Pętla for powyżej, działa na jednym rdzeniu, wystarczy przekształcić pętlę w funkcję i wykonać ją na wielu rdzeniach.
1 mnozenie <- function(i){
2 c[i]*2
3 }
4 library(multicore)
5
6 mclapply(1:10000000,mnozenie,mc.cores = 12)
7
Czym różni się jedna i druga operacja? Pętla wykonuje kroki po kolei 1,2,3,4,… itd., dopóki nie przejdzie przez wszystkie zadania, nie zatrzyma się. W przypadku przetwarzania wielordzeniowego, funkcja wykonuje po 12 kroków na raz. Ukończenie jednego z kroków spowoduje wejście kolejnego. W ten sposób dane są przetwarzane na bieżąco, a zwalniany rdzeń obliczeniowy automatycznie przetwarza kolejny wolny element funkcji. Im więcej rdzeni tym szybciej procesy zostaną ukończone. Dodatkowym atutem jest to, że „pętla” nie musi czekać na ukończenie wszystkich kroków, lecz po wykonaniu zwalnia się na przyjęcie kolejnych porcji danych. W przypadku powyższego kodu, mnożenie przetworzy się 12 razy szybciej, niż w standardowej pętli. Duży zbiór danych, liczący parę miliardów rekordów, przetworzy się w ten sposób znacznie szybciej.
Jak zapisywać duże zbiory danych?
Tutaj nie ma skutecznego sposobu na szybki zapis. Nie dlatego, że nie ma metod szybkiego zapisu BigData do plików, lecz wiąże się to z ograniczeniem systemowym. Zapis danych na Linuksie może się okazać zupełnie nieczytelny dla Windowsa i odwrotnie. Warto jednak zwrócić uwagę na kilka faktów. Jaki system docelowy będzie odbiorcą danych, czy dane mają być skompresowane, jak dane będą przechowywane?
Jeżeli dane chcemy zapisać szybko do plików tekstowych, w tym przypadku polecam paczkę readr, która w bardzo szybki sposób tworzy wielowymiarowe dane tekstowe. Taki typ danych jest najpopularniejszy i najłatwiejszy do przetworzenia na różnych systemach i programach. Pamiętajmy jednak o znaku końca linii, który kolokwialnie mówiąc służy za „enter”. System wie, kiedy pojawia się nowa linia. Inaczej wygląda to na systemie Linux i inaczej na systemie Windows. Pracując na R w systemie UNIXowym, funkcja write.table, pozwala na ustawienie parametru eol. Wpisanie wartości nr spowoduje zapis końca linii widoczny dla Windowsa, natomiast wpisując r, znak końca linii będzie widoczny dla Linuxa.
Dlaczego zwracam uwagę na taką rzecz? Zbiory danych, które zawierają parę milionów rekordów mogą zablokować program, którym będą wczytywane. Może się okazać także, że może być to przyczyną zawieszenia systemu i konieczności jego restartu.
Znak końca linii można podejrzeć w edytorze tekstowym Notepad++, tak jak widać to poniżej.
Oprócz tego typu informacji w zapisanych plikach, powinniśmy uwzględniać kodowanie znaków, ponieważ może to spowodować błędny odczyt/zapis znaków specjalnych.
Przy przetwarzaniu BigData powinny zostać uwzględnione wszystkie możliwe warianty, od budowy kodu, poprzez jego przyśpieszenie innymi językami programowania, aż po wykorzystanie wszystkich rdzeni i dostępnej pamięci RAM, a także tzw. partycji wymiany (zarezerwowane miejsce na dysku, uaktywnia się gdy pamięć ram jest zapełniona).
Moim zdaniem to podstawowa recepta na uniknięcie problemów związanych z bardzo dużymi zbiorami danych oraz recepta na przyśpieszenie pracy. W końcu czas to pieniądz.
Zdjęcie główne artykułu pochodzi z stocksnap.io.
Podobne artykuły
Podejście do zmniejszania kosztów przetwarzania danych na przykładzie Azure Databricks

Data management, data governance i data modernization - jak okiełznać proces zarządzania danymi w organizacji

Big Data - klęska urodzaju czy złoty Graal? Praktyczne metody wykorzystania potencjału danych
Dane 1,3 mln użytkowników Clubhouse do pobrania? To nie wyciek, to data scraping

Text clustering, czyli jak wyciągnąć realną wartość biznesową z milionów wiadomości tekstowych

Migracja do chmury? Wypełnij ankietę i sprawdź, jak wygląda ten proces u innych!

Analiza infrastruktury i danych IoT. Wybór bazy